甲型流感病毒(H1N2)亚型毒株血凝素和神经氨酸酶基因来源的研究
中华实验和临床病毒学杂志 2000年第1期第14卷 论著
作者:祁俊林 王敏 董婕 张烨 郭俊峰 郭元吉
单位:祁俊林(100052 北京,中国预防医学科学院病毒学研究所流感研究室);王敏(100052 北京,中国预防医学科学院病毒学研究所流感研究室);董婕(100052 北京,中国预防医学科学院病毒学研究所流感研究室);张烨(100052 北京,中国预防医学科学院病毒学研究所流感研究室);郭俊峰(100052 北京,中国预防医学科学院病毒学研究所流感研究室)
关键词: 正粘病毒A型,人;血凝素;神经氨酸酶;基因
【摘要】 目的 研究新分离到的H1N2亚型毒株血凝素(HA)和神经氨酸酶(NA)基因的来源。方法 病毒通过鸡胚增殖后提取其RNA,通过逆转录合成cDNA,经PCR扩增和产物纯化,用双脱氧链终止法进行核苷酸序列测定,并用MegAlign(1.03版)和Editseq(3.69版)软件进行种系发生学分析。结果 新分离到H1N2毒株HA1区氨基酸序列与A/PR/8/34(H1N1)和A/Guangdong/6/91(H1N1)毒株间的同源性分别为98.2%和99.4%;NA蛋白分子氨基酸序列与1995年的甲3(H3N2)亚型毒株间同源性高达99.1%。结论 新分离的H1N2毒株是A/PR/8/34(H1N1)类毒株与甲3(H3N2)亚型毒株通过基因重配而来。
Study on the origin of influenza A(H1N2) virus HA and NA genes
Qi Junlin, WANG Min, DONG Jie, et al.
(Institute of Virology, Chinese Academy of Preventive Medicine, Beijing 100052)
【Abstract】 Objective To determine the origins of the HA and NA genes of new subtype (H1N2) of influenza A virus. Methods Virus was amplified in embryonated chicken eggs, then virion RNA was transcribed into cDNA by reverse transcriptase, cDNA amplified by PCR, the products of PCR were purified. Afterward, RNA sequence analysis was performed by the dideoxynucleotide chain termination method, using synthetic oligodideoxynucleotide primers. Finally, phylogenetic analysis of the sequencing data was performed with MegAlign (Version 1.03) and Editseq (Version 3.69) softwares. Results The homology of amino acid sequences of protein molecules on HA1 domains when compared, with A/PR/8/34(H1N1) and A/Guangdong/6/91(H1N1) viruses was 98.2% and 99.4%, respectively. The homology of amino acid sequences of NA protein molecules between the novel reassortant and H1N1 virus was only 83.4%, but the homology of those between H1N2 strain and H3N2 virus circulating in men in 1995 was as high as 99.1%. Conclusion The new reassortant(H1N2) virus HA gene was derived from A/PR/8/34(H1N1) like virus, but its NA gene was derived from H3N2 virus circulating in men in 1995.
【Key words】 Influenza A virus,human; Haemagglutinin; Neuraminidase; Genes
甲型流感病毒曾造成多次世界性流感大流行,给人类带来了深重的灾难。但流感病毒大流行株起源至今尚未弄清,仍存在着种种不同的看法。有人认为,流感病毒大流行株是人与禽流感病毒通过基因重配而来[1]。1977年,H1N1亚型流感病毒在人群中消失20年后又重现[2],它的基因组与50年代在人群流行的H1N1亚型毒株极为相似[3],显然它的重现很难用重配学说来加以解释。然而,它的重现给了解不同亚型流感病毒是否能在人群中发生基因重配带来了良机。自1977年以来,人群中同时流行着两个亚型(H3N2和H1N1)毒株。而后,确实国内外学者相继报道了人群中分离出H3N2和H1N1病毒的基因重配株[4~9]。由于人群对这些重配株普遍存在有不同程度的免疫力,故它们均未能在人间造成流感流行。
1996年,我们在流感监测中又分离出了3株H1N2亚型流感病毒。出乎意料的是,它们的血凝素蛋白抗原性类似于A/PR/8/34(H1N1)毒株[10]。据我们所知,这样的重配株属国际上首次发现,这个发现对弄清流感病毒抗原性变异规律,尤其大流行株起源具有重要的意义。故有必要弄清新分离出H1N2亚型毒株的基因来源。下面报告其HA和NA基因来源的研究结果。
1 材料和方法
1.1 病毒 A/太铁/26/95(H1N2)、A/太铁/41/95(H1N2)和A/太铁/42/95(H1N2)系于1995年12月至1996年3月分别从12,12和36岁三名流感患者中分离出,经9~10 d龄鸡胚尿囊腔接种增殖,所收获的新鲜尿囊液为实验用病毒。
1.2 病毒粒RNA提取 按德国1995年出品的RNeasy RNA kit,QIAquickTM Handbook所提供的方法。
1.3 cDNA合成 聚合酶链式反应见文献[11]。
引物:用于HA基因,cDNA合成和PCR扩增及序列测定的有:6(5′ d-AAGCAGGGGAAAATA-AAA),282(5′d-CTGCTTCCAGTGAGAT CAT),505(5′d-TTTACAGAAATTTGCTATGG),792(5′d-ATAATATT TGAGGCAAATGGA),1193R(5′d-GTAATCCCGTTAATG GCA);用于NA基因,cDNA合成和PCR扩增及序列测定的有:14(5′d-GTGAAGA TGAATCCAAATCAA),106(5′d-GGTA-ACTACGTTAACATTGCA),372(5′d-GAGAACCTTATGTGTCATGCG),695(5′d-CAGGAGTCGG AATGCGTTTGT),823(5′d-GTCAGGAAGTGC-TCAGCA),1063(5′d-AGTGAAAGGCTGG-GCCTT),1420R(5′d-GCGAAAGCTTATATAGGC)。
所有引物由国家流感中心提供。35S-ATP,逆转录酶,PCR试剂盒,PCR产物纯化试剂盒及Sequence system试剂盒均由美国疾病控制中心提供。
1.4 PCR产物纯化 按德国出品的QIAquick PCR Purification kit所提供的方法。
1.5 核苷酸序列测定 采用双脱氧链终止法[12],基本步骤按USB公司DNA sequence试剂盒所提供的方法。
1.6 核苷酸序列分析 用DNA STAR公司出品的序列分析软件,MegAlign(1.03版)和Editseq(3.69版)。A/Beijing/262/95(H1N1)的序列由美国疾病控制中心提供,其它序列由国家流感中心提供。
2 结果
2.1 血凝素基因核苷酸序列测定和分析 由于流感病毒HA轻链(HA2)区很保守,而重链(HA1)区具有易变性。前言中已提到,新分离出的H1N2毒株HA蛋白的抗原性类似于A/PR/8/34(H1N1)病毒。故对三株H1N2病毒的HA1区基因核苷酸序列进行了测定,并将测定结果推导出氨基酸序列,然后,与一些甲1亚型毒株进行比较,见图1。

“-”为糖基化位点
1 3株H1N2毒株与A/PR/8/34(H1N1)类毒株,当时流行H1N1毒株及以往发现的H1N2毒株HA1区蛋白分子上氨基酸序列比较
Fig.1 Deduced amino acid sequences of the HA gene HA1 region of the three novel H1N2 viruses compared with those of H1N1 and H1N2 viruses
Note:The amino acid sequences of the HA1 protein molecule of all the comparing viruses compared were provided by the National Influenza Center of China. “-”means the essential glycosylation site
图1表明:测定和比较毒株HA1基因均编码328个氨基酸,有共同的6个糖基化位点,HA1与HA2连接处均为一个精氨酸(R);3株H1N2毒株HA1蛋白分子上见不到有氨基酸序列的差异;而H1N2与A/PR/8/34(H1N1)毒株间有6个位点不同,分别位于83(V-A),131(S-N),197(D-E),201(I-L),213(T-S)和262(R-W),其同源性为98.2%(322/328),然而,与A/Guangdong/6/91(H1N1)毒株间仅有两个差异:131(S-N)和262(R-W)。其同源性高达99.4%(326/328)。这些差异中除131和197分别位于A和B抗原决定簇区外,其余均位于非抗原决定簇区。
同时,图1还表明三株H1N2病毒HA1区氨基酸序列与当时人群中流行的甲1(H1N1)A/北京/262/95及以往发现的H1N2亚型毒株A/哈防/1/88的同源性仅分别为83.4%和84.0%。
2.2 神经氨酸酶基因核苷酸序列测定和分析 对3株H1N2病毒NA基因全序列进行测定并推导出其氨基酸序列。然后,与以往发现的H1N2亚型毒株A/Harbin/1/88和A/Harbin/12/92及1992~1995年的一些H3N2亚型毒株进行比较和分析,结果见图2。



“-”为糖基化位点
2 3株H1N2毒株与当时流行H3N2毒株及以往发现的H1N2毒株NA蛋白分子上氨基酸序列比较
Fig.2 Deduced amino acid sequences of the NA genes of the three novel H1N2 viruses compared
with those of previous H1N2 viruses and H3N2 viruses circulating in men recently
Note:the amino acid sequences of the NA protein molecule of previous H1N2 viruses and the H3N2 viruses were provided by the National Influenza Center of China. “-”means the glycosylation site
图2表明:测定和比较毒株NA基因均编码469个氨基酸,除两株旧的H1N2毒株仅有7个糖基化位点(少一个329~331位)外,其余的都有共同的8个糖基化位点。3株H1N2毒株NA蛋白分子上见不到有氨基酸序列的差异,而新发现H1N2与以往H1N2毒株A/Harbin/1/88、A/Harbin/12/92间分别差异31和732个氨基酸,但新H1N2与1995年流行的H3N2毒株间仅相差4~6个氨基酸。尤其与A/Shenzhen/272/95(H3N2)毒株仅差异4个氨基酸,两者之间的同源性高达99.1%(465/469氨基酸)。然而,与1992和1993年的H3N2毒株差异分别高达15和8个氨基酸。同源性达96.8%和98.3%。
2.3 HA和NA基因发生学分析
由于三株所分离出的H1N2毒株HA1和NA氨基酸序列完全相同,故下面图3和4中仅列出一株。HA基因发生学分析结果见图3。
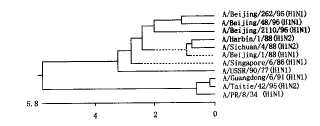
水平线代表毒株间不同核苷酸的数目,标尺代表毒株间突变的距离
3 11株甲型流感病毒(H1N1/H1N2)HA1基因序列的进化树
Fig.3 Phylogenetic tree for the HA1 genes of H1 subtype of influenza A viruses
Horizontal distance is proportional to the numbers of nucleotide differences between branch points. Vertical lines are for spacing. Scale is drawn to show the mutational distances between the viruses
图3结果清楚表明,A/Taitie/42/95(H1N2)毒株HA基因进化体系属A/PR/8/34(H1N1)这类毒株体系,而与其它H1N1和H1N2毒株体系不同。NA基因发生学分析结果见图4。
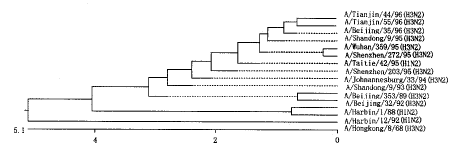
水平线代表毒株间不同核苷酸的数目,标尺代表毒株间突变的距离
4 15株甲型流感病毒(H3N2/H1N2)NA基因序列的进化树
Fig.4 Phylogenetic tree for 15 influenza A (H3N2 or H1N2) virus NA gene sequences
Horizontal lines are proportional to the numbers of nucleotide differences between branch points. Vertical lines are for spacing. Scale is drawn to show the mutational distances between the viruses
图4结果表明,A/Taitie/42/95毒株NA基因与1995年的H3N2毒株的NA基因最接近,与其它被比较的N2毒株有较大差异。
3 讨论
病毒粒HA1和NA基因核苷酸序列测定及其发生学分析进一步证实,3株太铁病毒的HA和NA基因分别为H1和N2亚型,并表明它们是A/PR/8/34(H1N1)类似毒株与1995年人群中流行的H3N2亚型毒株通过基因重配而来。它们显然不同于我们过去所发现的H1N2亚型毒株[8,9]。发现它们的意义也将会大大超过以往所发现的H1N2重配毒株。因它不仅进一步证实了不同亚型流感病毒能直接在人群中发生基因重配,而且指出了以往认为在人群中已消失的毒株,可能以人们不能测量的方式存在于人群中,在特定条件下,它们又会重新出现,甚至会再次在人群中造成流感流行,也就是说流感病毒大流行株出现不一定都与禽流感病毒有关。
众所周知,A/PR/8/34(H1N1)这类毒株,一般认为它于1946年已在人群中消失。因此,必然出现一个有趣的问题,A/PR/8/34(H1N1)这类毒株从何而来?为进一步排除它来自于实验室,除过去我们所提出的理由外[10],还进行了血清流行病学调查,结果在小于20岁人群中查到A/PR/8/34的特异抗体[13,14],加上本文图1结果,故我们认为:近期所发现的H1N2毒株来自实验室的可能性可完全加以排除。至今在我国人群仍存在有A/PR/8/34(H1N1)这类毒株的活动。至于A/PR/8/34(H1N1)和新分离出的H1N2毒株能否在人群中引起流感流行?这是人们关心和感兴趣的另一个问题。我们认为:这种可能性是存在的,必须给以足够的关注。
至于H1N2毒株其它基因节段的来源,我们正在抓紧研究之中。
基金项目:本课题为国家自然科学基金资助项目(39770038)
作者单位:郭元吉(100052 北京,中国预防医学科学院病毒学研究所流感研究室)
参考文献
1,Scholtissek C, Sohde W, Hoyningen V, et al. On the origin of the human influenza virus subtypes H2N2 and H3N2. Virology,1978,87:12-20.
2,Kung HC, Jen KF, Yuan WC, et al. Influenza in 1977:recurrence of influenza virus A subtype H1N1. Bull. WHO,1978,56:913-918.
3,Nakajima K, Desselberger V, Palese P. Recent human influenza A (H1N1) viruses are closely related genetically to strains isolated in 1950. Nature (London),1978,274:334-339.
4,Young JF, Peter P. Evolution of human influenza viruses in nature:recombination contributes to genetic variation of H1N1 strains. Proc Nathl Acad Sci U S A,1979,76:6547-6551.
5,Yamane N, Avikawa J, Odagiri T, et al. Isolation of three different influenza A viruses from an individual after probable double infection with H3N2 and H1N1 viruses. Jpn J Sci Biol,1978,31:431-434.
6,Kendal AP, Lee DT, Parish HS, et al. Laboratory based surveillance of influenza virus in the United States during the winter of 1977-1978. Ⅱ. Isolation of a mixture of A/Victoria and A/USSR-like viruses from a single person during an epidemic in Wyoming U S A,January, 1978.Am J of Epid,1979,110:462-468.
7,Nishikawa F, sugiyama T. Direct isolation of H1N2 recombinant virus from a throat swab of a patient simultaneously infected with H1N1 and H3N2 influenza viruses. J Clin Microbiol,1983,18:425-427.
8,郭元吉,徐西雁,Cox NJ,等.H1N2新亚型流感病毒的发现及其起源的调查.中华实验和临床病毒学杂志,1990,4:133-146.
9,郭元吉,郭忠明,王敏,等.从我国人群中再次分离到重分配的H1N2亚型流感病毒.中华实验和临床病毒学杂志,1993,7:121-126.
10,郭元吉,祁俊林,王敏,等.我国人群中一种新重配的H1N2亚型流感病毒株的发现.中华实验和临床病毒学杂志,1997,11:101-103.
11,Saiki PK,Delfand DH, Stattels A, et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science,1988,239:487-491.
12,Cox NJ, Kitame F, Klimov A, et al. Comparative studies of wild type and cold mutant (temperature-sensitive) influenza viruses: detection of mutation in all genes of the A/Ann Arbor/6/60(H2N2) mutant vaccine donor strain. Microbial Pathogenesis,1986,1:387-397.
13,郭元吉,王敏,程小雯.从人群中分离到一株A/PR/8/34(H1N1)类病毒.中华实验和临床病毒学杂志,1997,11:101-104.
14,郭元吉,祁俊林,王敏,等.A/PR/8/34(H1N1)病毒株血清流行病学调查.中华实验和临床病毒学杂志,1998,12(3):223-225.
(收稿日期:1998-12-01)